NHRI Communications

院務紀事
核心儀器設施中心淺談次世代核酸定序(NGS)技術與應用—16S菌種鑑定之新服務
NHRI to provide next generation sequencing service
 1984年開始的人類基因體計畫,以第一代Sanger定序方法耗時超過10年花費30億美元的資金,才完成整個人類基因體的定序。2007年次世代定序(next generation sequencing, NGS)技術問世,整體定序技術開始快速地演進與發展,大幅降低定序所需的成本和時間,至今單次輸出已可達540-600 Gb(Illumina system)的資料量,再結合生物資訊分析的技術,NGS已廣泛應用於各學術研究領域。
1984年開始的人類基因體計畫,以第一代Sanger定序方法耗時超過10年花費30億美元的資金,才完成整個人類基因體的定序。2007年次世代定序(next generation sequencing, NGS)技術問世,整體定序技術開始快速地演進與發展,大幅降低定序所需的成本和時間,至今單次輸出已可達540-600 Gb(Illumina system)的資料量,再結合生物資訊分析的技術,NGS已廣泛應用於各學術研究領域。目前NGS技術可應用於下列研究:
DNA level:
- 全基因體定序(whole genome sequencing)
- 新基因(genome de novo)
- 外顯子與目標區域(exome & target region)
- ChIP sequencing
- 轉錄體(Transcriptome)
- 微核糖核酸(miRNA)expression
- 表觀遺傳學(甲基化)((epigenetics (methylation))
- 菌相分析(metagenomics)
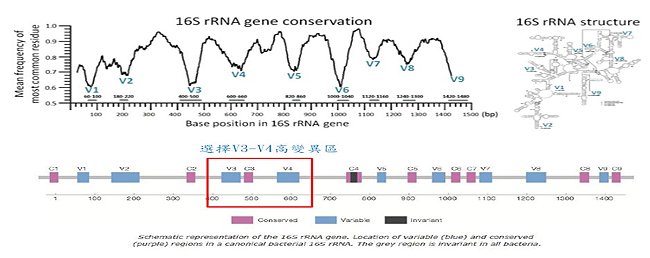
將來院內研究人員如欲使用核心實驗室所提供的16S菌種鑑定定序建庫(library)服務,樣本準備時須注意以下事項:
- DNA的量至少要有500 ng以上,須含大片段DNA(>10 kb),可自行以電泳圖先行判斷,並由核心實驗室進行最後的Bioanalyzer測定判別。有無大片段DNA將是影響建庫成功與否的重要關鍵。
- 有無RNA殘留。可利用NanoDrop(會測得DNA+RNA)與Qubit(單測DNA)來測量濃度數值,如差距太大表示有RNA 殘留,需再使用RNase處理檢體。
- 採集糞便檢體要注意即時低溫(冷藏或冷凍)保存,或使用糞便穩定採集管,以保留原始的菌相分析狀態。
《文/圖:核心儀器設施中心粘書瑋、蘇特立》