NHRI Communications

研究發展
次世代基因體定序資料分析之序列組裝整合工具
CISA: Contig Integrator for Sequence Assembly of Bacterial Genomes
 次世代基因體定序(Next Generation Sequencing, NGS)技術的快速發展,促使各研究領域積極利用次世代定序技術來進行基因解碼。因應分析NGS技術所產出的高通量之短序列,各種序列組裝工具(sequence assembler)也相繼發展,目的為加速完成全新(de novo)物種基因體序列。目前已有許多序列組裝工具能夠針對次世代基因體的序列加以組裝,然而各個基因體組裝的結果卻不盡相同,針對各個分歧的物種,沒有一個工具能夠擔保會提供最佳的組裝結果,因此本院群體健康科學研究所生物統計與生物資訊研究組廖玉潔助研究員的研究團隊開發了一個序列組裝整合工具「CISA: Contig Integrator for Sequence Assembly of Bacterial Genomes」,目的在整合各種不同組裝工具所產出的序列,找出相互吻合的序列片段,同時排除歧異度過高的序列,最後提供一組整合後的基因體序列;經過嚴謹地驗證程序檢驗,研究團隊所開發之工具確實能夠有效地整合並提升細菌基因體序列的完整性。
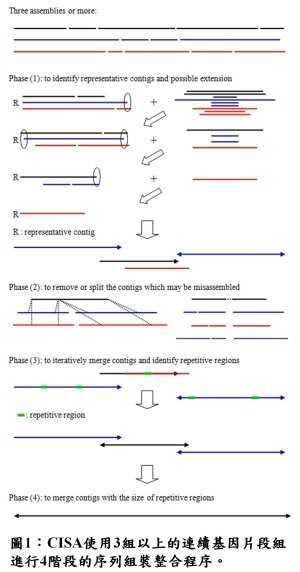
次世代基因體定序(Next Generation Sequencing, NGS)技術的快速發展,促使各研究領域積極利用次世代定序技術來進行基因解碼。因應分析NGS技術所產出的高通量之短序列,各種序列組裝工具(sequence assembler)也相繼發展,目的為加速完成全新(de novo)物種基因體序列。目前已有許多序列組裝工具能夠針對次世代基因體的序列加以組裝,然而各個基因體組裝的結果卻不盡相同,針對各個分歧的物種,沒有一個工具能夠擔保會提供最佳的組裝結果,因此本院群體健康科學研究所生物統計與生物資訊研究組廖玉潔助研究員的研究團隊開發了一個序列組裝整合工具「CISA: Contig Integrator for Sequence Assembly of Bacterial Genomes」,目的在整合各種不同組裝工具所產出的序列,找出相互吻合的序列片段,同時排除歧異度過高的序列,最後提供一組整合後的基因體序列;經過嚴謹地驗證程序檢驗,研究團隊所開發之工具確實能夠有效地整合並提升細菌基因體序列的完整性。有鑒於使用不同的序列組裝工具,所獲得的連續基因片段(contig)之完整度與正確度常有部分出入,研究團隊發展的序列組裝整合工具CISA將依序進行4個步驟(圖1),來提高序列組裝之完整度。(1)找出並延伸代表基因片段組(representative contig):使用3組以上,由不同組裝工具,或相同組裝工具使用不同參數,所得到的連續基因片段組(assemblies);依序選出當中最長的代表片段,並做可能的延伸。(2)偵測出連續基因片段中可能組裝錯誤(misassembled)的序列,並將之移除。(3)將末端重疊30%以上的基因片段相互連接,此步驟重複執行直至無法再進行片段連接。(4)進行末端連結時同時估計基因片段組中重複性序列(repetitive regions)的最大長度,再利用此長度為依據進行後續末端連結與否的判斷條件,此步驟重複執行直至無法再進行片段連接。
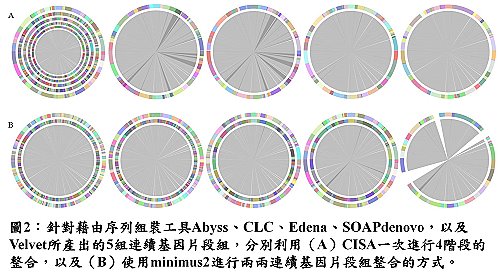
CISA分別針對3種物種E. coli、S. aureus以及H. volcanni之次世代序列資料進行分析,整合序列組裝工具包括Abyss、Edena、SOAPdenovo,以及Velvet等所產出的結果,再與每個單一組裝工具所產出的連續基因片段組比較,結果皆顯示出CISA不但能夠有效地提高基因體的完整度(較高的N50以及較少的連續基因片段數),同時還能維持基因體組裝的正確度(具有較多的完整編碼序列(intact coding sequence)),此外,CISA藉由同時分析多組之連續基因片段組以產出整合的基因體序列(圖2A)的方式,與一次只能整合兩組基因體序列的工具,例如minimus2以疊代方式所產生之整合結果(圖2B)比較,顯示出使用CISA能夠確保提供完整之基因體序列。此項研究工作已發表於PLoS ONE期刊(PLoS ONE 8(3):e60843)。
《文/圖:群體健康科學研究所生物統計與生物資訊研究組廖玉潔助研究員、林介華研究助理、林信宏研究助理》